文章目录
RNA-FISH定位
RNA FISH即RNA荧光原位杂交,是一种利用荧光标记的核酸探针对细胞或组织中的RNA进行定性、半定量或相对定位分析的技术。
1.RNA FISH 技术指南

2.单细胞基因表达检测-RNA-FISH和流式FlowRNA
目前,聚合酶链式反应(Polymerase Chain Reaction,PCR)和免疫组化(Immuno Histo Chemistry,IHC)是进行生物标志物(DNA/RNA和蛋白)检测的最常用的方法。但它们都有其各自的技术局限性:
聚合酶链式反应(PCR)用于检测DNA和RNA虽然灵敏度高,但在提取核酸的过程中,RNA部分降解、低峰度基因表达的RNA不可避免的会出现丢失,导致无法检测。而且PCR只是检测了细胞内核酸的平均水平,无法对核酸进行单细胞水平的定位检测。
免疫组化(IHC)主要是利用抗原与抗体特异性结合原理,通过化学反应使标记抗体的显色剂(荧光素、酶、金属离子、同位素)显色来确定组织细胞内的抗原(多肽和蛋白质),并对其进行定位、定性及定量的研究。由于需要特异性抗体,因此对于一些没有特异性抗体的蛋白,采用免疫组化检测显然不可能;即使可以制备抗体,也需要花费几个月的时间来完成。对于一些低浓度表达的分泌性蛋白信号,免疫组化的敏感性也比较差。另外,对于保守性比较高的基因家族,往往由于抗体的特异性较差而无法检测出基因家族内蛋白的差异性。
对于基因及功能的检测,一般通过检测DNA和蛋白来实现。然而,不是所有的RNA都编码蛋白,部分RNA可能只是参与了基因调控和其它广泛的细胞活动。而且RNA表达的变化可能不是源于DNA的变化,也不一定引起蛋白质水平变化(如长链非编码RNA(LncRNA))。因此,检测RNA最好的方法是能进行原位检测,精确定位其在细胞和组织微环境中位置并进行定量分析。
RNA原位杂交(ISH)技术的出现解决了PCR无法对细胞或组织内靶RNA定位,免疫组化(IHC)非特异性结合和传统原位杂交中可能出现的一系列问题。
ViewRNA、Primeflow RNA技术由双“Z”探针设计,
(1)其具有高度特异性、极高的信噪比的特点;
(2)同时能够在单细胞单分子水平同时定位、定量分析多个RNA的表达;
(3)可在保持组织和细胞形态的条件下,单细胞单分子水平对细胞内RNA进行定位、定量的分析方法;
(4)该技术解决了PCR无法定位,免疫组化(IHC)非特异性结合和传统原位杂交中可能出现的一系列问题;并且可以与免疫组化(IHC)结合应用,检测RNA与蛋白的表达情况。

RNA-FISH可用于多种类型样本检测分析:
- 缺乏有效抗体的蛋白抗原检测(如:斑马鱼、肿瘤干细胞CD133检测);
- 稀有珍贵样本中基因表达分析;
- 同源基因家族筛查;
- LncRNA(长链非编码RNA)检测;
- 样本中部分降解RNA的检测;
- 嵌合体分析;
- RNA单细胞单分子水平定位、定量分析;
- 测序后的基因表达结果验证;
- 多基因表达的定量、定位分析;
- 感染性RNA病毒检测等。
联系我们(xieyl@novobiotec.com, shangzh@novobiotec.com)获取FISH-Flow Nature Protocol:
3.PinPoRNA
原位杂交(in situ hybridization)技术已经被广泛应用于检测目标蛋白或DNA分子在细胞或组织切片上的位置和数量,但对于大部分目标RNA分子的检测,传统方法由于特异性和灵敏度无法兼顾,通常无法得到好的检测结果。PinpoRNA™原位杂交技术是由广东品博易视生物科技有限公司自主专利研发的RNA原位杂交检测试剂。与传统的RNA原位杂交相比,PinpoRNA™属于新一代RNA原位杂交技术,是唯一可以更好地替代进口产品得到可媲美结果的国产试剂。
PinpoRNA™原位检测原理
PinpoRNA™试剂盒利用创新的短片段探针(20-50 bp)设计(专利号:CN202110581853.9)和特别的核酸蛋白级联信号放大系统(专利号:CN202110575231.5),兼顾解决了特异性和灵敏度问题,可以在组织切片上检测到低至1-10拷贝的目标RNA分子。只要目标RNA分子序列已知且长于100碱基,我们即可设计相应的探针进行检测。整个实验大约需时6-7小时。其检测原理是:
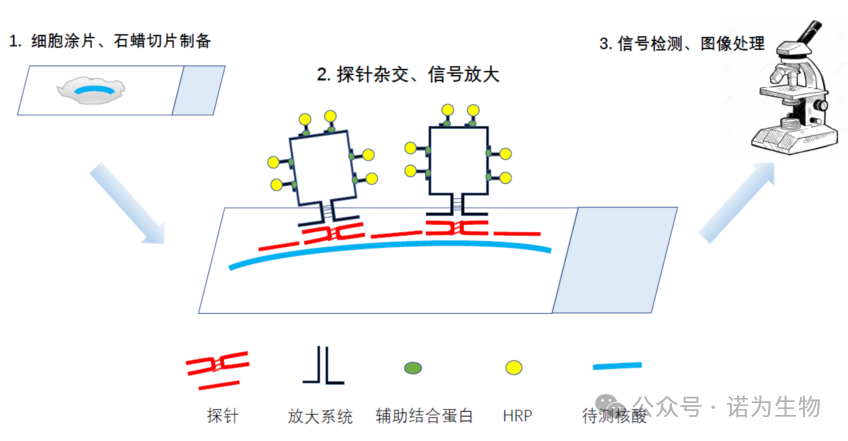
PinpoRNA™原位杂交试剂盒无需配套硬件及设备,操作简单,大幅降低了科研及临床用户的使用门槛和成本,相比国外竞争品牌,操作更简单,信噪比更高,更具经济效益。
PinpoRNA™原位检测特点
- 经济高效易用---产品性价比高,货期短,步骤更易操作,兼容各类国产杂交仪和实验器械,实验结果同比进口试剂。
- 应用广泛---使用PinpoRNA™技术,靶点RNA为大于100碱基的特异序列,即可进行探针设计(无设计费)。因而可以用于几乎所有物种,所有组织及所有基因的检测。
- 高特异高灵敏---专利的成组式探针设计保证了检测的特异性,同时提高了杂交效率。全新的信号放大体系使用更少的步骤实现同等信号放大,可在单细胞水平定量定位胞内RNA分子。
- 多通道多靶点检测---pinpoRNA有不同系列的自动或手动检测产品,可适用于普通光学显微镜,荧光显微镜,共聚焦显微镜,高通量全自动细胞扫描仪等在组织和各种细胞上观测目标RNA分子。
PinpoRNA™探针
PinpoRNA™ 通过每组探针 2-4 条同时杂交于指定区域实现检测,而这几条探针同时结合于非特异位点的可能性小于百万分之一。相比传统的原位杂交使用一段较长的探针(至少几百碱基),直接杂交于待测核酸,再通过该探针上的地高辛或荧光标记观察信号相比,PinpoRNA™探针对位点的变异敏感,可以区分同家族基因,检测灵敏度高、特异性强。
PinpoRNA™试剂盒
PinpoRNA™试剂盒有化学染色和荧光染色,化学染色有红色和棕色,荧光染色有单色荧光试剂盒、双色荧光试剂盒和三色荧光试剂盒,可以实现在一张片子上同时检测三个靶标。也可以实现RNA和蛋白的共定位。
4.PrimeFlow RNA
PrimeFlow RNA分析将专利的寡核苷酸探针组设计与分支DNA (bDNA)信号放大技术相结合,利用流式细胞术分析RNA转录本。bDNA技术提供了独特的RNA检测和信号放大方法,通过放大报告基团信号而不是靶序列(如PCR),有助于获得较PCR的分析一致性更高的结果。在PrimeFlow RNA分析中,靶点特异性的探针组包含20至40个可与目标RNA转录本杂交的寡核苷酸对(图1)。信号放大的原理是,相邻的寡核苷酸对与bDNA结构(包括信号预放大探针、放大探针和荧光标记的探针)特异性地杂交(图2),获得极佳的特异性、低背景和高信噪比。
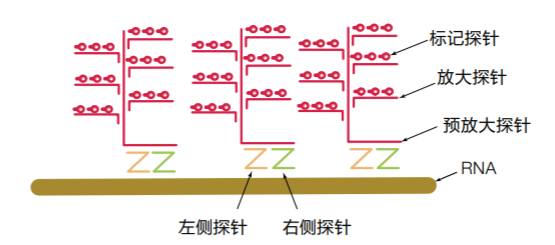
图1. PrimeFlow RNA分析中bDNA结构。
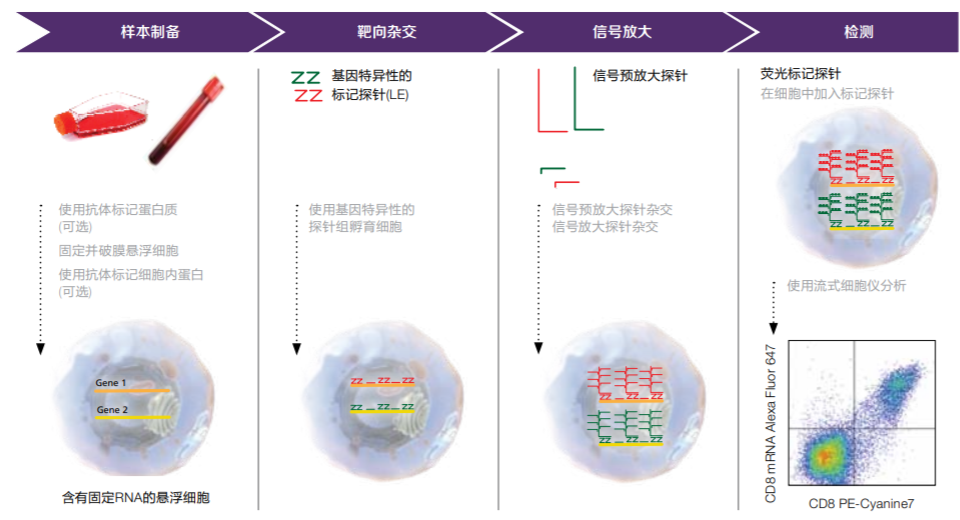
图2. PrimeFlow RNA分析工作流程。该分析工作流程包括下列几个步骤:抗体染色;固定和破膜,包括细胞内染色(如需要);然后是与包含20–40个寡核苷酸对的靶点特异性探针组的靶向杂交。
PrimeFlow RNA分析基于成熟且有大量文献支撑的ViewRNA技术开发而成,它利用显微镜分析细胞和组织内的RNA。改进后的PrimeFlow RNA分析将成对寡核苷酸探针设计与bDNA信号放大技术还有流式细胞术相结合,可以在标准流式细胞仪上可靠地检测单细胞中多达4种RNA转录本。
主要特点:
- 单细胞水平的基因表达异质性
- 单细胞中的RNA和蛋白质动力学
- 检测细胞亚群中的非编码RNA
- 评估感染细胞中的病毒RNA
- 抗体选择有限时分析mRNA表达水平
- 同时分析多达4种RNA转录本
- 流式细胞术检测microRNA (miRNA)
结果展示
1.同一细胞中的RNA和蛋白质动力学
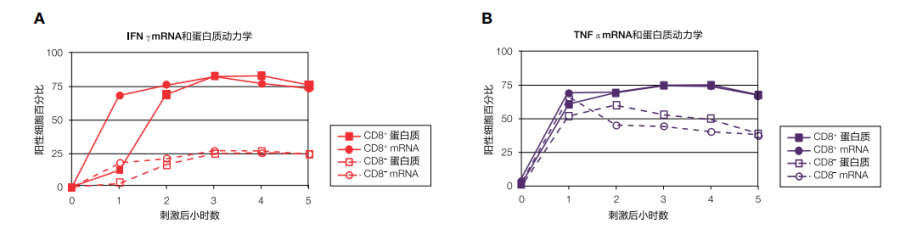
图3. 利用PrimeFlow RNA分析检测IFNγ和TNFα转录和翻译动力学。使用Invitrogen™ eBioscience™细胞刺激混合物(含蛋白转运抑制剂) (货号:00-4975)刺激正常人外周血单核细胞0–5小时。使用PrimeFlow RNA分析,固定并破膜处理细胞,然后进行CD8、IFNγ和TNFα抗体的细胞内染色。随后,细胞经过一系列的杂交步骤,标记IFNγ和TNFα mRNA。淋巴细胞设门内的活的CD8+和CD8–细胞用于分析。
2. 单细胞水平的基因表达异质性
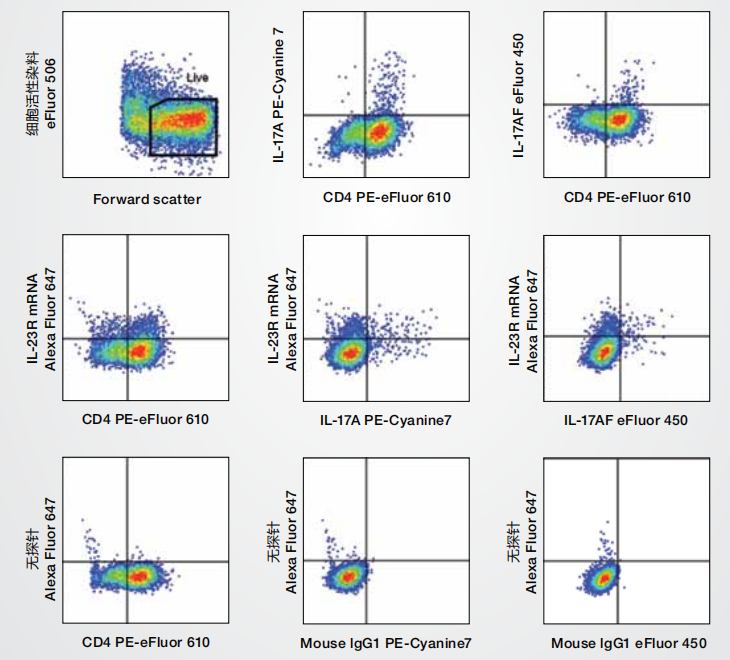
图4. 利用PrimeFlow RNA分析检测Th17细胞亚群中的IL-23R mRNA表达。正常人外周血细胞在Th17极化条件下培养3天,然后使用eBioscience细胞刺激混合物(含蛋白转运抑制剂) (货号:00-4975)重新刺激5小时。使用Invitrogen™ eBioscience™ Fixable Viablity Dye eFluor™ 506 (货号:65-0866)标记细胞,使用PrimeFlow RNA分析缓冲液固定并破膜,然后用抗CD4 PE-eFluor™ 610、抗IL-17A PE-Cyanine7和抗IL-17AF eFluor™ 450进行细胞内染色。随后,细胞经过一系列的杂交步骤,标记IL-23R mRNA。
3.评估感染细胞中的病毒RNA
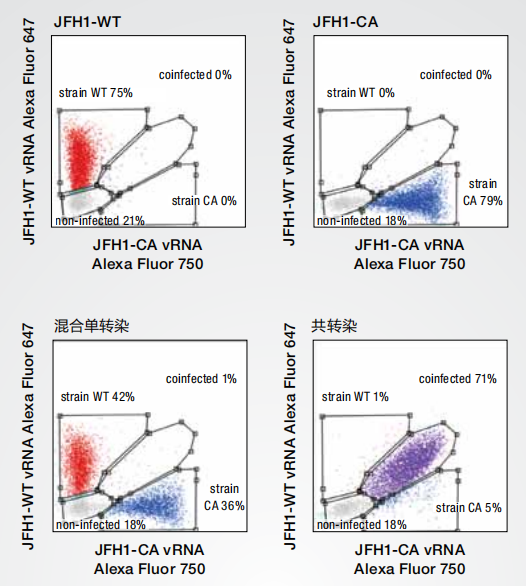
图5. 单细胞共感染病毒RNA原理验证。* 肝细胞感染JFH1-WT菌株 (左上) 或 JFH1-CA (右上)。感染后,将分别感染上述两种病毒的单感染肝细胞混合(左下),或者肝细胞同时感染JFH1-WT和JFH1-CA (右下)。使用Invitrogen™ QuantiGene™ FlowRNA分析,对肝细胞中的菌株特异性病毒RNA进行分析。
5.Stellaris RNA Fish
Stellaris® RNA FISH 是一种检测细胞,组织切片,动物整体,组织或器官中单个 RNA 分子或 RNA 集群的方法。RNA-FISH 可以对空间上分离的 RNAs 进行定位、定量,由于探针可设计多种颜色,因而其可对多基因表达的相互影响进行定位、定量及Flow-FISH检测。
Stellaris® RNA FISH 是一种用于检测固定细胞或者组织中单个或成簇 RNA 的新型技术。多条特异性探针具有相似的稳定性,与目标单分子 RNA 杂交后可以产生荧光信号。LGC Biosearch Technologies 为客户提供基于网站免费的且易于操作的 Stellaris RNA FISH 探针设计软件,software for Stellaris RNA FISH Probe design https://www.biosearchtech.com/Account/Login?return=/stellaris-designer。仅需要输入一条序列,软件即可以输出一组至多由 48 条探针组成的探针集,这些探针具有最佳结合特性。
通常探针数量不少于 25 就可靠地检测。对于大部分长链且特异的目标 RNA,较容易达到 25 条。但是针对一些特别短的或者重复序列的 RNA 就比较困难,一开始得到 25 条及以上探针的可能性较小。这种情况下可以通过改变设计软件的一些参数来增加探针的条数。虽然并不是所有的目标 RNA 均能与 Stellaris RNA FISH 技术兼容,利用以下设计策略可以成功实现对于一些具有挑战性的目标 RNA 进行检测。
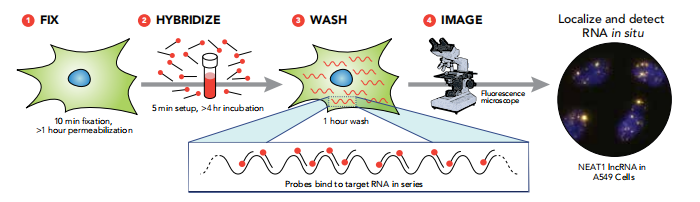
步骤A:样品准备制备组织样品切片或细胞玻片,加酒精透膜,无需蛋白酶处理!注:制备样品时应尽量避免 RNA 被 RNA 酶污染而导致其降解。不同样品类型制备的具体方案请与我们联系。
步骤B:杂交探针可在37°C恒温环境下杂交4-16小时。注:探针分为目录探针和定制探针,其中定制的探针集可采用我们的在线 Stellaris® 探针设计软件(www.biosearchtech.com/stellarisdesigner)设计。
步骤C:洗涤样品去除杂交缓冲液,加洗涤缓冲液短时间孵育,以去除多余的探针。此步骤需要1-1.5小时。
步骤D:成像RNA在荧光显微镜下呈现点状信号,选择合适的荧光显微镜光源和镜头成像。
储存条件
Stellaris RNA FISH 探针
干燥运输,2–8°C 避光储存。溶解后应尽量减少反复冻融次数。溶解后的探针短期内用完,建议在 2–8°C 条
件下避光储存,且最多不超过一个月。超过一个月储存时,务必分装并在 -15 – -30°C 条件下避光储存。
Stellaris RNA FISH 杂交缓冲液
Stellaris RNA FISH 杂交缓冲液在 2–8°C 条件下储存。
Stellaris RNA FISH 缓冲洗液 A 和缓冲洗液 B
Stellaris RNA FISH 缓冲洗液 A 和 B 在室温条件下储存。
其他必备试剂和设备
试剂耗材
a) TE 缓冲液(10 mM Tris-HCl, 1 mM EDTA, pH 8.0)
b) 甲醇
c) 冰醋酸
d) 37% 甲醛溶液
e) 乙醇,分子生物学级别
f) 10X 磷酸盐缓冲液(PBS),无 RNA 酶
g) 无核酸酶水
h) 去离子甲酰胺
i) Stellaris RNA FISH 杂交缓冲液(LGC Biosearch Technologies 公司 Cat# SMF-HB1-10)
j) Stellaris RNA FISH 缓冲洗液 A(LGC Biosearch Technologies 公司 Cat# SMF-WA1-60)
k) Stellaris RNA FISH 缓冲洗液 B(LGC Biosearch Technologies 公司 Cat# SMF-WB1-20)
l) 4',6- 二脒基 -2- 苯基吲哚(DAPI)
m) Vectashield® 防荧光淬灭封片剂(Vector Laboratories 公司 Cat #H-1000)
n) 具有 #0 或 #1 厚度盖玻片的 96 孔玻璃底细胞培养板 *
o) 矿物油
p) 无 RNA 酶的耗材,如移液器吸头
q) 37°C 恒温孵育箱
6.Nature Protocols手把手教您用流式多重分析单细胞的RNA和蛋白
常规的FISH技术可以揭示qPCR忽略掉的细胞间异质性的问题,但是对显微镜的依赖限制了它的取样能力(通常<100细胞),这往往会丢失掉一些低丰度细胞的重要信息,比如Circulating Tumor Cells (CTC,循环肿瘤细胞)! 想要高通量的进行单个细胞水平的基因表达检测需要依靠新技术Flow-FISH。
Nature Protocols的大神们发明了一套基于流式细胞仪的FISH技术,可以进行多重的、高通量的单细胞水平基因表达分析,包括RNA和蛋白两个层面的表达分析。这一新技术即是FISH-Flow(文献链接:http://www.nature.com/articles/nprot.2017.039)联系我们索要全文:xieyl@novobiotec.com, shangzh@novobiotec.com 或010-57438396
简单来说,作者利用LGC Biosearch 的Stellaris FISH针对待测的RNA设计高达48根探针(可自选荧光标记的类型),然后按照作者文章里的步骤处理细胞,就可以通过流式细胞仪同时观察细胞的RNA和蛋白表达情况。相比较其它RNA-FISH实验步骤,我们提供的Stellaris FISH探针实验操作步骤简捷是这一技术成功的关键。
|
|
|||||
|
Figure Comparison of mRNA expression analysis by microscopy and FISH-Flow. (a) Schematic representation of sm-FISH probes tiled along the length of an mRNA target. Binding of multiple fluorescently labeled probes to each mRNA molecule results in fluorescent signals that are bright enough to enable detection by flow cytometry and by fluorescence microscopy.(b) Representative microscopy images of HeLa cells after sm-FISH using c-fos probes.(c) Scatter plots of HeLa cells after hybridization with probes against c-fos mRNA, GFP mRNA (a negative control) and GAPDH mRNA (a positive control). Gates were set separately, based on no-serum controls in each case. A total of 50,000 events per sample were analyzed. |
联系我们(010-57438396或xieyl@nwbiotec.com, heyy@nwbiotec.com)获取FISH-Flow Nature Protocol:
7.用于癌症基因表达研究的Stellaris® RNA FISH 探针
基因表达谱较为复杂,而且在组织、细胞甚至单个核的不同区域也有着较大的差异,因此需要先进的分析方法。RNA FISH 技术可以实现单分子转录本RNA的可视化与定量化,而且在固定细胞或者完整组织样品中观察分析疾病特异性表达的变化方面有独特的能力。
诺为生物提供的Stellaris RNA FISH探针可以对形态完整的细胞、组织切片或者wholemount组织中的RNA 进行定量和定位分析。Stellaris RNA FISH 探针集由多个针对一个目标RNA 的寡核苷酸组成。荧光标记的探针结合在靶转录本上,使得单个目标RNA 分子产生点状信号进而被检测。
我们提供免费的在线探针设计软件,针对目标RNA 设计最佳的Stellaris RNA FISH 探针。Custom Stellaris RNA FISH 探针组是由至多48 个寡核苷酸组成的混合物,每个寡核苷酸带有荧光基团标记。此外我们也提供DesignReady 探针组,经过专业的设计以及严格的生物信息学分析以确保其特异性。这些定制的探针集包含众多生物模型的癌症特异性的靶标。更多信息和实验方法请登录网站(www.biosearchtech.com/stellaris) 查询。探针设计请咨询:010-57438396, E-mail: xieyl@novobiotec.com或shangzh@novobiotec.com

|
人腺癌(SK-BR-3) 细胞中,Stellaris RNA FISH 和免疫荧光方法共定位表达分析: ERBB2 mRNA(绿色),ERBB2 (HER2) 蛋白质(品红色)和 DNA(红色)(AV Orjalo Biosearch Tech.) |
|
ADAM17 |
EGFR |
HOTAIR |
MYC |
AKT1 |
ERBB2&3 (HER2) |
HOXA5 |
NCOA3 |
|
AR |
ERG |
JAG1 |
PCA3 |
BCL2 |
EZH2 |
JUN |
PIK3CA |
|
BRAF |
FOS |
KIT |
MTOR |
BRCA1&2 |
GREB1 |
KLK3 |
TERT |
|
CDK4 |
H19 |
MALAT1 |
TERC |
CTNNB1 |
HIF1A |
MKI67 |
TP53 |

Cell, Nature和Science发表的文献中超过33%引用Stellaris RNA-FISH方法:
Post-transcriptional Modifications Contribute to the Upregulation of Cyclin D2 in Multiple Myeloma.
Misiewicz-Krzeminska et al. Clinical Cancer Research. 2016. url: https://dx.doi.org/10.1158/1078-0432.CCR-14-2796
G1/S Inhibitors and the SWI/SNF Complex Control Cell-Cycle Exit during Muscle Differentiation.
Ruijtenberg et al. Cell. 2015. url: https://dx.doi.org/10.1016/j.cell.2015.06.013
Genome-wide profiling of p53-regulated enhancer RNAs uncovers a subset of enhancers controlled by a lncRNA.
Leveille et al. Nature Communications. 2015. url: https://dx.doi.org/10.1038/ncomms7520
Niche appropriation by Drosophila intestinal stem cell tumours.
Patel et al. Nature Cell Biology. 2015. url: https://dx.doi.org/10.1038/ncb3214
FuseFISH: robust detection of transcribed gene fusions in single cells.
Semrau et al. Cell reports. 2014. url: https://dx.doi.org/10.1016/j.celrep.2013.12.002
Proteostatic Control of Telomerase Function through TRiC-Mediated Folding of TCAB1.
Freund et al. Cell. 2014. url: https://dx.doi.org/10.1016/j.cell.2014.10.059
Single-molecule transcript counting of stem-cell markers in the mouse intesting.
Itzkovitz et al. Nature Cell Biology. 2011. url: https://dx.doi.org/10.1038/ncb2384
8.RNA ISH实验中石蜡切片样品的预处理
固定样品 → 组织块的切片 → 固定组织切片 → 切片脱水 → 画阻水圈 → 样品预处理
一、固定样品
样品剖分后,立即放入 10% 中性福尔马林(NBF),在室温(RT)下固定组织16-32小时。根据组织类型和大小,固定时间有所不同。
二、样本的脱水、包埋和切片
1.用 1X PBS 清洗样品。
2.使用乙醇进行逐级脱水,然后使用二甲苯对样品进行透明。
3.采用标准流程将样品包埋在石蜡中。
注:包埋的样品在15-20°C下干燥保存,若保存时间超过1年,为确保RNA的质量,建议在2-8°C下保存。
4.根据需要修剪石蜡块,用切片机将包埋的组织切成5 ± 1 μm 的切片。石蜡切片放入40-45°C的水浴中,将切片贴附到SUPERFROST. PLUS载玻片上。
5.在室温下过夜风干载玻片。
注:请使用 3 个月以内制备的组织切片。请在室温下使用干燥剂储存切片。
三、烤片
在干燥箱中于60°C下烤片1 小时。
注:烤片完成后立即使用,或在室温下使用干燥剂储存。储存时间不超过 1 周,储存过久可能会导致样品 RNA 降解。
四、FFPE 切片脱石蜡
1.在通风橱中准备4个清洗皿:
在两个清洗皿中加入约 200 mL 新鲜的二甲苯。
在两个染色皿中加入约 200 mL 新鲜的 100% 乙醇。
2.将载玻片放入载玻片架,并在通风橱中将其浸入第一个盛有二甲苯的皿中。
3.将二甲苯中的载玻片于室温下孵育 5 分钟。不时地上下移动载玻片架。
4.从第一个盛有二甲苯的皿中取出载玻片架,然后立即放入第二个盛有二甲苯的皿中。
5.重复步骤3。
6.从第二个盛有二甲苯的皿中取出载玻片架,然后立即放入盛有100%乙醇的皿中。
7.将 100% 乙醇中的载玻片于室温下孵育1分钟,孵育时需不停地摇动。
8.使用新鲜的 100% 乙醇重复步骤 6 和 7。
9.从载玻片架上取下载玻片,切片朝上置于吸水纸上。在室温下风干载玻片5分钟至完全干燥。
五、组织切片预处理
1.把载玻片浸入70% 乙醇中,室温孵育2分钟。
注:任何时候不要让细胞变干。总是使用足够的溶液浸没所有的细胞。
2.去掉70% 乙醇,更换为50% 乙醇。室温孵育2分钟。
3.去掉50% 乙醇,更换为1×PBS。室温孵育10分钟。
4、双氧水处理10分钟。
5、画阻水圈。
6、蛋白酶Plus处理约 30-60分钟。
9.新鲜冰冻组织的样本制备和预处理方法
固定样品 → 组织块的切片 → 固定组织切片 → 切片脱水 → 画阻水圈 → 样品预处理
一、固定样品
1. 取出组织,切割组织块使其符合冰冻切片用包埋模型。
注:固定时间少于16小时或长于32 小时将影响分析结果。
2. 在获得组织后的5分钟之内在干冰或液氮中冰冻组织。
3. 使用低温包埋剂(OCT)包埋冰冻组织:
a.加两滴OCT到一个冰冻切片包埋模型中。
b.在适合切片的正确方向把冰冻组织放到OCT上。
c.加更多的OCT来填满冰冻切片包埋模型。不要产生任何气泡。
d.用手术钳夹住冰冻切片包埋模型保持在液氮表面或干冰/液氮冷却的异戊烷的表面,或将其放到干冰上。
4. 切片前,冰冻组织块保存于-80℃条件下的密封容器中。
注:包埋的组织可以保存至少3个月。
二、组织块的切片
1. 在低温恒温器中于-20℃下平衡组织块~约1小时。
2. 切10-20 μm厚 的切片,粘附到SuperFrost. Plus载玻片上。
3. 在-20℃放置切片至干燥。
4. 把切片保存在载玻片盒中,用铝箔或自封袋密封-80℃保存直至使用。
注:切片至多可以保存3个月。
三、固定切片
1. 在一个Tissue Tek®染色皿中将200毫升10% NBF(新鲜制备的或溶于1× PBS的 4% PFA)降温至4℃。
2. 从-80°C取出载玻片,并放置到Tissue Tek®载玻片架上。
3. 立即将载玻片浸在预冷的固定液中,4℃下固定15分钟。
注:已储存超过6个月、或暴露于空气中超过一周、或反复使用的福尔马林都可能导致组织固定不佳。
四、切片脱水
1. 在Tissue Tek®染色皿中分别配制200 mL 50% 乙醇、200 mL 70% 乙醇和600 mL 100% 乙醇。
2. 把载玻片放入50% 乙醇中,室温下(RT)下放置5分钟。
3. 把载玻片放入70% 乙醇中,室温下放置5分钟。
4. 把载玻片放入100% 乙醇中,室温下放置5分钟。
5. 用新鲜的乙醇重复步骤4。
注:载玻片在-20°C 条件下于100% 乙醇中保存不超过1周。保存时间延长可能降解样本中RNA。
五、画阻水圈
1. 从100%乙醇中取出载玻片,放到吸水纸上,切片面朝上,室温下空气中干燥5分钟。
2. 使用Immedge阻水笔按照以下模板在每个切片周围画阻水圈 2-4 次。
六、样品预处理
1. 加入蛋白酶IV 完全覆盖每一个组织。
2. 室温孵育30 分钟。
3. 每次处理一个载玻片,快速去除过量液体,然后将载玻片放入1×PBS 洗涤。
4. 在染色皿中上下移动载玻片架以洗涤载玻片,用1×PBS 重复洗涤一次。
10.RNA ISH实验中固定后冰冻切片的样本的制备和预处理
第1 部分:组织切片的制备
A. 固定样本
1. 如果需要,用新鲜制备的含4% 多聚甲醛(PFA)的1×PBS 灌注组织,或直接转到步骤2。
2. 解剖组织并放到新鲜制备的4%PFA 中,4℃ 下固定24 小时。
B. 冷冻组织
1. 4℃下把组织浸入含10% 蔗糖的1×PBS 中,直到组织沉到容器底部(脑组织大约18 小时)。
2. 在含20% 蔗糖的1×PBS 中重复这一步骤,之后是在含30% 蔗糖的1×PBS 中,每次都要等组织沉到容器底部。
3. 在OCT 包埋介质中用干冰或液氮冷冻组织,并将其放在一个密封的容器中储存于-80℃冰箱中。
C. 制备切片
1. 在组织切片前,在低温恒温器中,于-20℃下平衡组织块至少1 小时。
2. 以7-15 μm 厚度切组织块,将切片粘附到SuperFrost. Plus 载玻片(Fisher Scientific # 12-550-15)上。
3. 在-20℃空气干燥载玻片20 分钟,或如果不立即使用载玻片,于-80℃保存载玻片< 3 个月。
4. 载玻片置于Tissue-Tek® 载玻片架上,用200 mL 1×PBS 清洗载玻片5 分钟,上下移动载玻片架去除OCT。
第2 部分:组织预处理
1、加双氧水去除内源性过氧化氢酶,室温(RT)孵 育10分钟。使用足够的溶液完全覆盖切片。用蒸馏 水清洗切片。
2、画阻水圈。
3、加蛋白酶处理。
11.RNA ISH实验中PBMC和悬浮细胞样本的制备和预处理方法
PBMC 纯化
1. 转移 5 mL 血液到 15 mL 离心管中。
2. 小心地把 5 mL 血液加在 Histopaque®1077 溶液上面。
3. 室温下用水平转子以 800 RCF 离心 20 分钟(使用最小的加速 / 制动)。
4. 用移液器或吸取装置小心地移去上层相(血浆相),在 PBMC 层上面留下 ~ 约 0.5 厘米液体。然后加入细胞稀释液稀释到适当浓度。
5. 室温下以 250 RCF 离心 10 分钟(使用最大的加速/ 制动)。
6. 吸弃上清液而不搅动细胞团,留下约 5 mL 的液体。
7. 上下吹吸 10 次重悬细胞团,然后转移到一个新的15 mL 离心管中。
8. 用新鲜的稀释液重悬细胞,使细胞损失降到最低。
9. 室温下以 250 RCF 离心 10 分钟(使用最大加速 /制动)。
10. 在不碰到细胞团的前提下吸出上清液,留下尽可能少的液体。

悬浮细胞的收集
1. 在一个 50ml 离心管中, 室温下以 250 RCF离心 10分钟收集细胞。
2. 吸去上清,不要搅动细胞。
3.40 mL 1× PBS 重悬细胞,然后在室温下以 250RCF 离心。
4. 在不碰到细胞团的前提下吸去上清,留下尽可能少的液体。
细胞固定
1. 用 5 mL PBMC- 固定液(10% NBF)重悬细胞,轻轻地上下吹吸 10 次完全吹散细胞团。
2. 在 37° C 水浴锅中孵育 1 小时。
固定后的洗涤和保存
1.250 RCF 离心 10 分钟。
2. 吸去上清液,不要搅动细胞团。
3. 用 10 mL PBMC- 洗涤液重悬细胞,250 RCF 离心10 分钟。
4. 用 10 mL PBMC- 保存液(70% EtOH)重悬细胞,上下吹吸 10 次完全吹散细胞团。
5. 室温下孵育 10 分钟,转移到 4° C。
注:细胞可以保存在 PBMC- 保存液(70% EtOH)中,于 4° C 下可保存 7 天。
玻片制备
1. 用 PBMC- 保存液调整细胞密度到 1×10e6 细胞 /mL。
2. 吹吸混合均匀,转移 1ml 细胞悬液到每一个预组装的细胞离心装置中。
注: 这里描述的细胞密度和体积是基于使用 8ml 腔室的 Hettich 细胞甩片机。如果使用其他甩片机,请调整细胞密度和体积以便在甩片后获得单细胞层。
3. 在 800 RCF 下甩片 20 分钟。
4. 用移液器小心地吸掉全部上清液,拆分甩片装置。
5. 室温下空气干燥载玻片 20 分钟。
6. 将载玻片浸到 50% 乙醇中, 室温孵育 5 分钟。
7. 去掉 50% 乙醇,用 70% 乙醇代替, 室温孵育 5 分钟。
8.去掉70%乙醇,用100%乙醇代替, 室温孵育5分钟。
9. 去掉 100% 乙醇,用新鲜 100% 乙醇代替, 室温孵育 5 分钟。
注: 载玻片可以于 -20° C 下,在 100% 乙醇中保存至 1 个月。
12.RNA ISH中培养的贴壁细胞的样本制备与预处理
细胞培养
1. 在细胞固定前一天,把细胞接种到含生长培养基的腔室培养载玻片上,接种细胞密度以固定时达到 80-90% 为宜。
细胞固定
1. 弃掉生长培养基,拆卸腔室。
2. 把玻片浸入含有 1×PBS 的染色皿中。
重要! 任何时候不要让细胞变干。总是使用足够的溶液浸没所有的细胞。
3. 去掉 1×PBS,加入 10% 中性缓冲福尔马林(NBF)。室温(RT) 下孵育 30 分钟。
4. 去掉 NBF,用 1×PBS 轻轻冲洗玻片,重复两次。
脱水和保存细胞
1. 去掉最后洗涤的 1×PBS,更换为 50 mL 50% 乙醇。RT 孵育 5 分钟。
2. 去掉 50% 乙醇,更换为 50 mL 70% 乙醇。 RT 孵育 5 分钟。
3. 去掉 70% 乙醇,更换为 50 mL 100% 乙醇。 RT孵育 5 分钟。
4. 去掉 100% 乙醇,更换为新鲜的 100% 乙醇。 RT孵育 10 分钟。
注: 细胞玻片在 100% 乙醇中、 -20℃条件下可保存长达 6 个月。
细胞再水合
1. 把载玻片浸入 70% 乙醇中, RT 孵育 2 分钟。
重要! 任何时候不要让细胞变干。总是使用足够的溶液浸没所有的细胞。
2. 去掉 70% 乙醇,更换为 50% 乙醇。 RT 孵育 2 分钟。
3. 去掉 50% 乙醇,更换为 1×PBS。 RT 孵育 10 分钟。
建立一个疏水圈
1. 在腔室载玻片上的每一个孔 / 圈周围用 Immedge ™疏水性屏障笔画 2-4 次,让疏水圈完全干透 ~1 分钟。
注: 在此步骤中不要让细胞变干。如果细胞太干就把载玻片放回到 1×PBS 中。
2. 在一个染色皿中用 1×PBS 洗载玻片。
蛋白酶透膜
1、加蛋白酶处理玻片。
2、用PBS清洗玻片。
13.RNA原位杂交实验中常见问题答疑
1、RNA检测技术越来越多,该如何选择?
答:RNA检测原理主要是通过设计与靶标互补配对的探针达到检测目标RNA的目的,不同技术的关键在于探针的设计原理,不同的探针类型适用的片段大小也有差异。并且RNA检测结果的好坏,除探针因素影响外,还与检测样本类型、荧光或可见光选择、显微镜等有关,具体细节可与我们技术沟通,联系方式请查看官网首页。
2、探针信号放大技术放大原理是什么?

通过碱基互补配对的原理对探针进行信号放大(如图所示)。
3、靶标没有信号怎么办?
答:靶标没有信号的影响因素很多,如固定时间、透膜方法、杂交时间等因素都会影响信号。首先在检测时要选择阳性对照,可以检测实验过程样本、试剂及操作是否有问题,然后再进行后续实验细节的分析。
4、对照探针有种属之分吗?
答:阳性对照一般是选用看家基因,不同种属相同基因是有差别的,因此选用的对照探针需要区分种属。
