文章目录
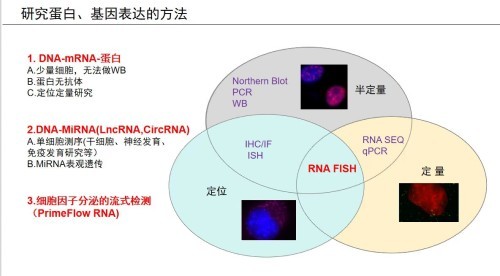
单细胞单分子RNA定位、定量分析---RNA Fish
RNA FISH即RNA荧光原位杂交,是一种利用荧光标记的核酸探针对细胞或组织中的RNA进行定性、半定量或相对定位分析的技术。
1. RNA Fish概述
聚合酶链式反应(Polymerase Chain Reaction,PCR)和免疫组化(Immuno Histo Chemistry,IHC)是进行生物标志物(DNA/RNA和蛋白)检测的最常用的方法。但它们都有其各自的技术局限性:
聚合酶链式反应(PCR)用于检测DNA和RNA虽然灵敏度高,但在提取核酸的过程中,RNA部分降解、低峰度基因表达的RNA不可避免的会出现丢失,导致无法检测。而且PCR只是检测了细胞内核酸的平均水平,无法对核酸进行单细胞水平的定位检测。
免疫组化(IHC)主要是利用抗原与抗体特异性结合原理,通过化学反应使标记抗体的显色剂(荧光素、酶、金属离子、同位素)显色来确定组织细胞内的抗原(多肽和蛋白质),并对其进行定位、定性及定量的研究。由于需要特异性抗体,因此对于一些没有特异性抗体的蛋白,采用免疫组化检测显然不可能;即使可以制备抗体,也需要花费几个月的时间来完成。对于一些低浓度表达的分泌性蛋白信号,免疫组化的敏感性也比较差。另外,对于保守性比较高的基因家族,往往由于抗体的特异性较差而无法检测出基因家族内蛋白的差异性。
对于基因及功能的检测,一般通过检测DNA和蛋白来实现。然而,不是所有的RNA都编码蛋白,部分RNA可能只是参与了基因调控和其它广泛的细胞活动。而且RNA表达的变化可能不是源于DNA的变化,也不一定引起蛋白质水平变化(如长链非编码RNA(LncRNA))。
基于以上目前实验方法的局限性,检测RNA一种不可或缺的检测技术就是能进行原位检测,精确定位其在细胞和组织微环境中位置并进行定量分析。
RNA原位杂交(ISH)技术的出现解决了PCR无法对细胞或组织内靶RNA定位,免疫组化(IHC)非特异性结合和传统原位杂交中可能出现的一系列问题。
RNA-FISH和RNA-ISH原位杂交技术有如下有点:
(1)其具有高度特异性、极高的信噪比的特点;
(2)同时能够在单细胞单分子水平同时定位、定量分析多个RNA的表达;
(3)可在保持组织和细胞形态的条件下,单细胞单分子水平对细胞内RNA进行定位、定量的分析方法;
(4)该技术解决了PCR无法定位,免疫组化(IHC)非特异性结合和传统原位杂交中可能出现的一系列问题;并且可以与免疫组化(IHC)结合应用,检测RNA与蛋白的表达情况。
 |
 |
2. PinPoRNA
原位杂交(in situ hybridization)技术已经被广泛应用于检测目标蛋白或DNA分子在细胞或组织切片上的位置和数量,但对于大部分目标RNA分子的检测,传统方法由于特异性和灵敏度无法兼顾,通常无法得到好的检测结果。PinpoRNA™原位杂交技术是由广东品博易视生物科技有限公司自主专利研发的RNA原位杂交检测试剂。与传统的RNA原位杂交相比,PinpoRNA™属于新一代RNA原位杂交技术,是唯一可以更好地替代进口产品得到可媲美结果的国产试剂。
PinpoRNA™原位检测原理
PinpoRNA™试剂盒利用创新的短片段探针(20-50 bp)设计(专利号:CN202110581853.9)和特别的核酸蛋白级联信号放大系统(专利号:CN202110575231.5),兼顾解决了特异性和灵敏度问题,可以在组织切片上检测到低至1-10拷贝的目标RNA分子。只要目标RNA分子序列已知且长于100碱基,我们即可设计相应的探针进行检测。整个实验大约需时6-7小时。其检测原理是:
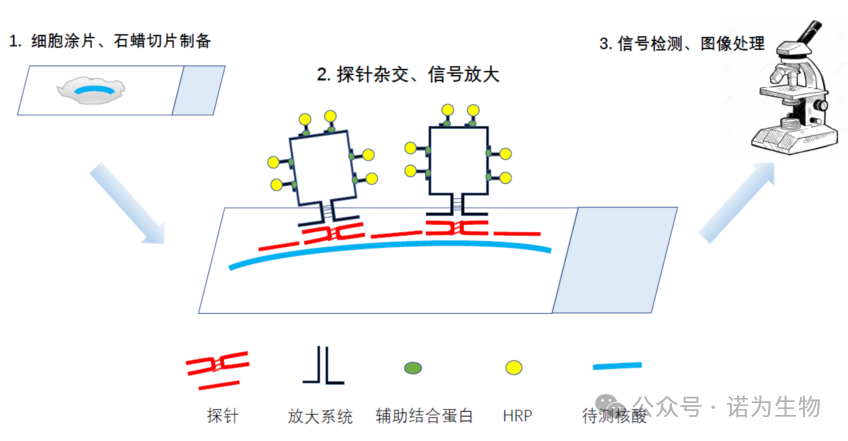
PinpoRNA™原位杂交试剂盒无需配套硬件及设备,操作简单,大幅降低了科研及临床用户的使用门槛和成本,相比国外竞争品牌,操作更简单,信噪比更高,更具经济效益。
PinpoRNA™原位检测特点
- 经济高效易用---产品性价比高,货期短,步骤更易操作,兼容各类国产杂交仪和实验器械,实验结果同比进口试剂。
- 应用广泛---使用PinpoRNA™技术,靶点RNA为大于100碱基的特异序列,即可进行探针设计(无设计费)。因而可以用于几乎所有物种,所有组织及所有基因的检测。
- 高特异高灵敏---专利的成组式探针设计保证了检测的特异性,同时提高了杂交效率。全新的信号放大体系使用更少的步骤实现同等信号放大,可在单细胞水平定量定位胞内RNA分子。
- 多通道多靶点检测---pinpoRNA有不同系列的自动或手动检测产品,可适用于普通光学显微镜,荧光显微镜,共聚焦显微镜,高通量全自动细胞扫描仪等在组织和各种细胞上观测目标RNA分子。
PinpoRNA™探针
PinpoRNA™ 通过每组探针 2-4 条同时杂交于指定区域实现检测,而这几条探针同时结合于非特异位点的可能性小于百万分之一。相比传统的原位杂交使用一段较长的探针(至少几百碱基),直接杂交于待测核酸,再通过该探针上的地高辛或荧光标记观察信号相比,PinpoRNA™探针对位点的变异敏感,可以区分同家族基因,检测灵敏度高、特异性强。
PinpoRNA™试剂盒
PinpoRNA™试剂盒有化学染色和荧光染色,化学染色有红色和棕色,荧光染色有单色荧光试剂盒、双色荧光试剂盒和三色荧光试剂盒,可以实现在一张片子上同时检测三个靶标。也可以实现RNA和蛋白的共定位。
3. PrimeFlow RNA
PrimeFlow RNA分析将专利的寡核苷酸探针组设计与分支DNA (bDNA)信号放大技术相结合,利用流式细胞术分析RNA转录本。bDNA技术提供了独特的RNA检测和信号放大方法,通过放大报告基团信号而不是靶序列(如PCR),有助于获得较PCR的分析一致性更高的结果。在PrimeFlow RNA分析中,靶点特异性的探针组包含20至40个可与目标RNA转录本杂交的寡核苷酸对(图1)。信号放大的原理是,相邻的寡核苷酸对与bDNA结构(包括信号预放大探针、放大探针和荧光标记的探针)特异性地杂交(图2),获得极佳的特异性、低背景和高信噪比。
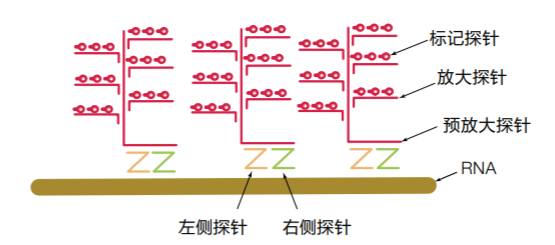
图1. PrimeFlow RNA分析中bDNA结构。
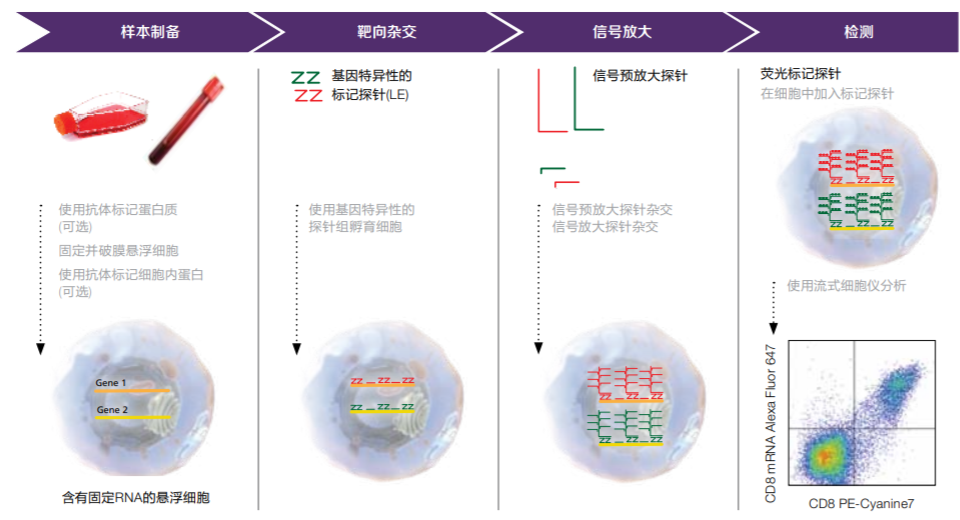
图2. PrimeFlow RNA分析工作流程。该分析工作流程包括下列几个步骤:抗体染色;固定和破膜,包括细胞内染色(如需要);然后是与包含20–40个寡核苷酸对的靶点特异性探针组的靶向杂交。
PrimeFlow RNA分析基于成熟且有大量文献支撑的ViewRNA技术开发而成,它利用显微镜分析细胞和组织内的RNA。改进后的PrimeFlow RNA分析将成对寡核苷酸探针设计与bDNA信号放大技术还有流式细胞术相结合,可以在标准流式细胞仪上可靠地检测单细胞中多达4种RNA转录本。
主要特点:
- 单细胞水平的基因表达异质性
- 单细胞中的RNA和蛋白质动力学
- 检测细胞亚群中的非编码RNA
- 评估感染细胞中的病毒RNA
- 抗体选择有限时分析mRNA表达水平
- 同时分析多达4种RNA转录本
- 流式细胞术检测microRNA (miRNA)
结果展示
1.同一细胞中的RNA和蛋白质动力学
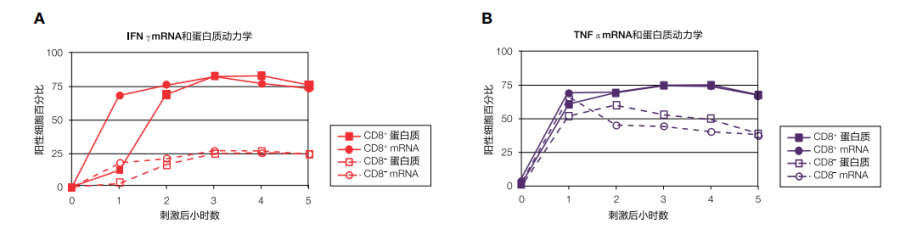
图3. 利用PrimeFlow RNA分析检测IFNγ和TNFα转录和翻译动力学。使用Invitrogen™ eBioscience™细胞刺激混合物(含蛋白转运抑制剂) (货号:00-4975)刺激正常人外周血单核细胞0–5小时。使用PrimeFlow RNA分析,固定并破膜处理细胞,然后进行CD8、IFNγ和TNFα抗体的细胞内染色。随后,细胞经过一系列的杂交步骤,标记IFNγ和TNFα mRNA。淋巴细胞设门内的活的CD8+和CD8–细胞用于分析。
2. 单细胞水平的基因表达异质性
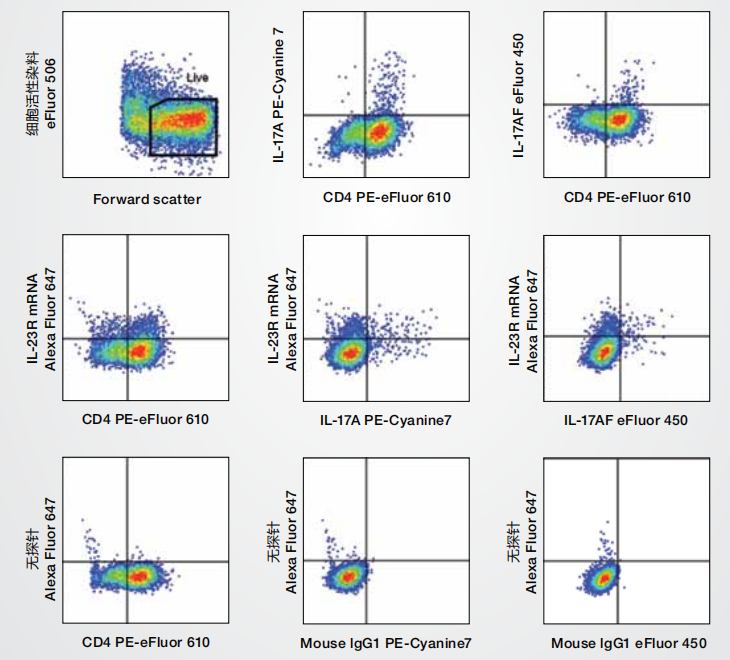
图4. 利用PrimeFlow RNA分析检测Th17细胞亚群中的IL-23R mRNA表达。正常人外周血细胞在Th17极化条件下培养3天,然后使用eBioscience细胞刺激混合物(含蛋白转运抑制剂) (货号:00-4975)重新刺激5小时。使用Invitrogen™ eBioscience™ Fixable Viablity Dye eFluor™ 506 (货号:65-0866)标记细胞,使用PrimeFlow RNA分析缓冲液固定并破膜,然后用抗CD4 PE-eFluor™ 610、抗IL-17A PE-Cyanine7和抗IL-17AF eFluor™ 450进行细胞内染色。随后,细胞经过一系列的杂交步骤,标记IL-23R mRNA。
3.评估感染细胞中的病毒RNA
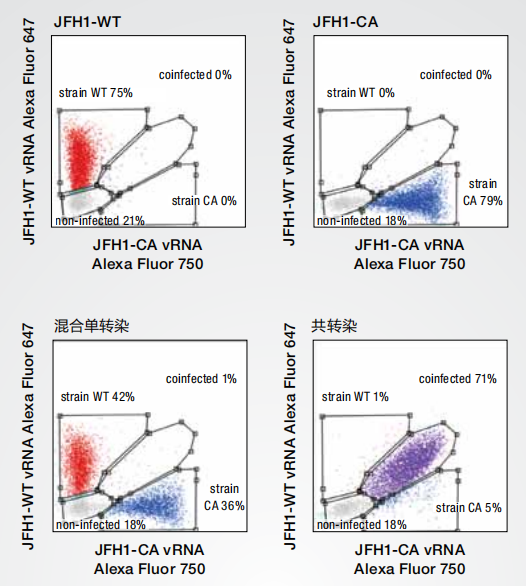
图5. 单细胞共感染病毒RNA原理验证。* 肝细胞感染JFH1-WT菌株 (左上) 或 JFH1-CA (右上)。感染后,将分别感染上述两种病毒的单感染肝细胞混合(左下),或者肝细胞同时感染JFH1-WT和JFH1-CA (右下)。使用Invitrogen™ QuantiGene™ FlowRNA分析,对肝细胞中的菌株特异性病毒RNA进行分析。
4. Stellaris RNA Fish
Stellaris® RNA FISH 是一种检测细胞,组织切片,动物整体,组织或器官中单个 RNA 分子或 RNA 集群的方法。RNA-FISH 可以对空间上分离的 RNAs 进行定位、定量,由于探针可设计多种颜色,因而其可对多基因表达的相互影响进行定位、定量及Flow-FISH检测。
Stellaris® RNA FISH 是一种用于检测固定细胞或者组织中单个或成簇 RNA 的新型技术。多条特异性探针具有相似的稳定性,与目标单分子 RNA 杂交后可以产生荧光信号。LGC Biosearch Technologies 为客户提供基于网站免费的且易于操作的 Stellaris RNA FISH 探针设计软件,software for Stellaris RNA FISH Probe design https://www.biosearchtech.com/Account/Login?return=/stellaris-designer。仅需要输入一条序列,软件即可以输出一组至多由 48 条探针组成的探针集,这些探针具有最佳结合特性。
通常探针数量不少于 25 就可靠地检测。对于大部分长链且特异的目标 RNA,较容易达到 25 条。但是针对一些特别短的或者重复序列的 RNA 就比较困难,一开始得到 25 条及以上探针的可能性较小。这种情况下可以通过改变设计软件的一些参数来增加探针的条数。虽然并不是所有的目标 RNA 均能与 Stellaris RNA FISH 技术兼容,利用以下设计策略可以成功实现对于一些具有挑战性的目标 RNA 进行检测。
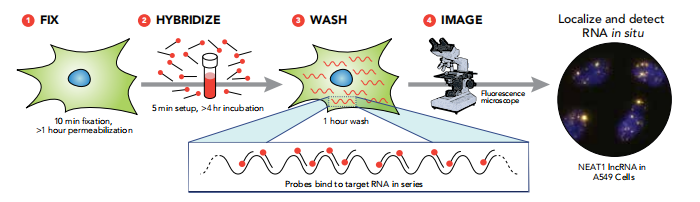
步骤A:样品准备制备组织样品切片或细胞玻片,加酒精透膜,无需蛋白酶处理!注:制备样品时应尽量避免 RNA 被 RNA 酶污染而导致其降解。不同样品类型制备的具体方案请与我们联系。
步骤B:杂交探针可在37°C恒温环境下杂交4-16小时。注:探针分为目录探针和定制探针,其中定制的探针集可采用我们的在线 Stellaris® 探针设计软件(www.biosearchtech.com/stellarisdesigner)设计。
步骤C:洗涤样品去除杂交缓冲液,加洗涤缓冲液短时间孵育,以去除多余的探针。此步骤需要1-1.5小时。
步骤D:成像RNA在荧光显微镜下呈现点状信号,选择合适的荧光显微镜光源和镜头成像。
储存条件
Stellaris RNA FISH 探针
干燥运输,2–8°C 避光储存。溶解后应尽量减少反复冻融次数。溶解后的探针短期内用完,建议在 2–8°C 条
件下避光储存,且最多不超过一个月。超过一个月储存时,务必分装并在 -15 – -30°C 条件下避光储存。
Stellaris RNA FISH 杂交缓冲液
Stellaris RNA FISH 杂交缓冲液在 2–8°C 条件下储存。
Stellaris RNA FISH 缓冲洗液 A 和缓冲洗液 B
Stellaris RNA FISH 缓冲洗液 A 和 B 在室温条件下储存。
其他必备试剂和设备
试剂耗材
a) TE 缓冲液(10 mM Tris-HCl, 1 mM EDTA, pH 8.0)
b) 甲醇
c) 冰醋酸
d) 37% 甲醛溶液
e) 乙醇,分子生物学级别
f) 10X 磷酸盐缓冲液(PBS),无 RNA 酶
g) 无核酸酶水
h) 去离子甲酰胺
i) Stellaris RNA FISH 杂交缓冲液(LGC Biosearch Technologies 公司 Cat# SMF-HB1-10)
j) Stellaris RNA FISH 缓冲洗液 A(LGC Biosearch Technologies 公司 Cat# SMF-WA1-60)
k) Stellaris RNA FISH 缓冲洗液 B(LGC Biosearch Technologies 公司 Cat# SMF-WB1-20)
l) 4',6- 二脒基 -2- 苯基吲哚(DAPI)
m) Vectashield® 防荧光淬灭封片剂(Vector Laboratories 公司 Cat #H-1000)
n) 具有 #0 或 #1 厚度盖玻片的 96 孔玻璃底细胞培养板 *
o) 矿物油
p) 无 RNA 酶的耗材,如移液器吸头
q) 37°C 恒温孵育箱